

Como
leer desde Excel.
- En excel nombrar las celdas correspondientes con un nombre, por ejemplo:
area
- En la primera linea de los datos renombrados debe estar la identificacion
de las columnas
- Grabar el archivo excel en un folder con el nombre, por ejemplo:
datos y cerrar el archivo.
- En R realizar las siguientes tareas:
- Instalar la libreria RODBC del CRAN
- Copiar el archivo correct rodbc
en el folder donde estan sus datos
- Cargar la libreria RODBC, que debe haber instalado en R:
>library(RODBC)
- Ejecutar el siguiente script con la orden:
>source("correct rodbc.R")
- continuar con las siguientes instrucciones:
>canal <- odbcConnectExcel("datos.xls")
>datos <- sqlFetch(canal, "area")
>odbcCloseAll()
AMMI.
- Realiza el analisis de variancia del analisis
combinado genotipo y ambiente, el analisis de las componentes principales,
su importancia y
significacion y finalmente realiza el Biplot o Triplot segun la importancia
de las componentes principales.
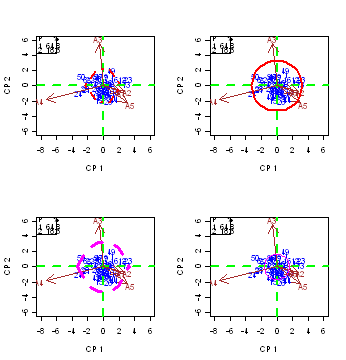 |
library(agricolae) # ver AMMI data(sinRepAmmi) REP <-3; MSerror <- 93.2422 # Realiza el analisis AMMI y el biplot par(mfrow=c(2,2),cex=0.5) model<-AMMI(ENV, GEN,
REP, YLD, MSerror,xlim=c(-8,6),ylim=c(-6,6)) model<-AMMI(ENV, GEN,
REP, YLD, MSerror,xlim=c(-8,6),ylim=c(-6,6)) model<-AMMI(ENV, GEN, REP, YLD,
MSerror,xlim=c(-8,6),ylim=c(-6,6)) model<-AMMI(ENV, GEN, REP, YLD, MSerror,xlim=c(-8,6),ylim=c(-6,6))AMMI.contour(model,distance=0.5,shape=20,col="magenta",lwd=1) |
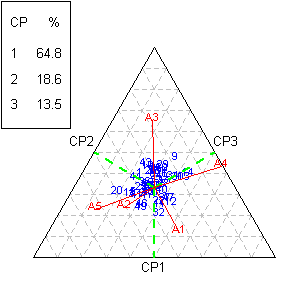 |
# Realiza el analisis
AMMI y el triplot library(klaR)
|